2025年4月14日,华中科技大学生命科学与技术学院宁康教授团队在国际权威期刊《Nucleic Acids Research》发表研究论文,创新性的推出了用于生物合成基因簇(Biosynthetic Gene Clusters, BGCs)识别的人工智能模型——BGC-Prophet。该工具在全球首次实现对超过8.5万个微生物基因组中百万级BGCs的系统性预测和注释,为合成生物学与新药研发提供了全新解决方案。
微生物基因组中的“语言密码”
生物合成基因簇(BGCs)是指一组在基因组中相邻排列、协同指导天然产物合成的功能模块,是抗生素、抗癌药物等活性物质的重要来源。传统的BGC识别依赖规则驱动的方法,面临识别范围有限、计算效率低等问题。
BGC-Prophet以自然语言处理中的Transformer架构为核心,将“语言模型”引入微生物基因组分析,将基因视作语义单元,实现对BGC的高效识别与分类。
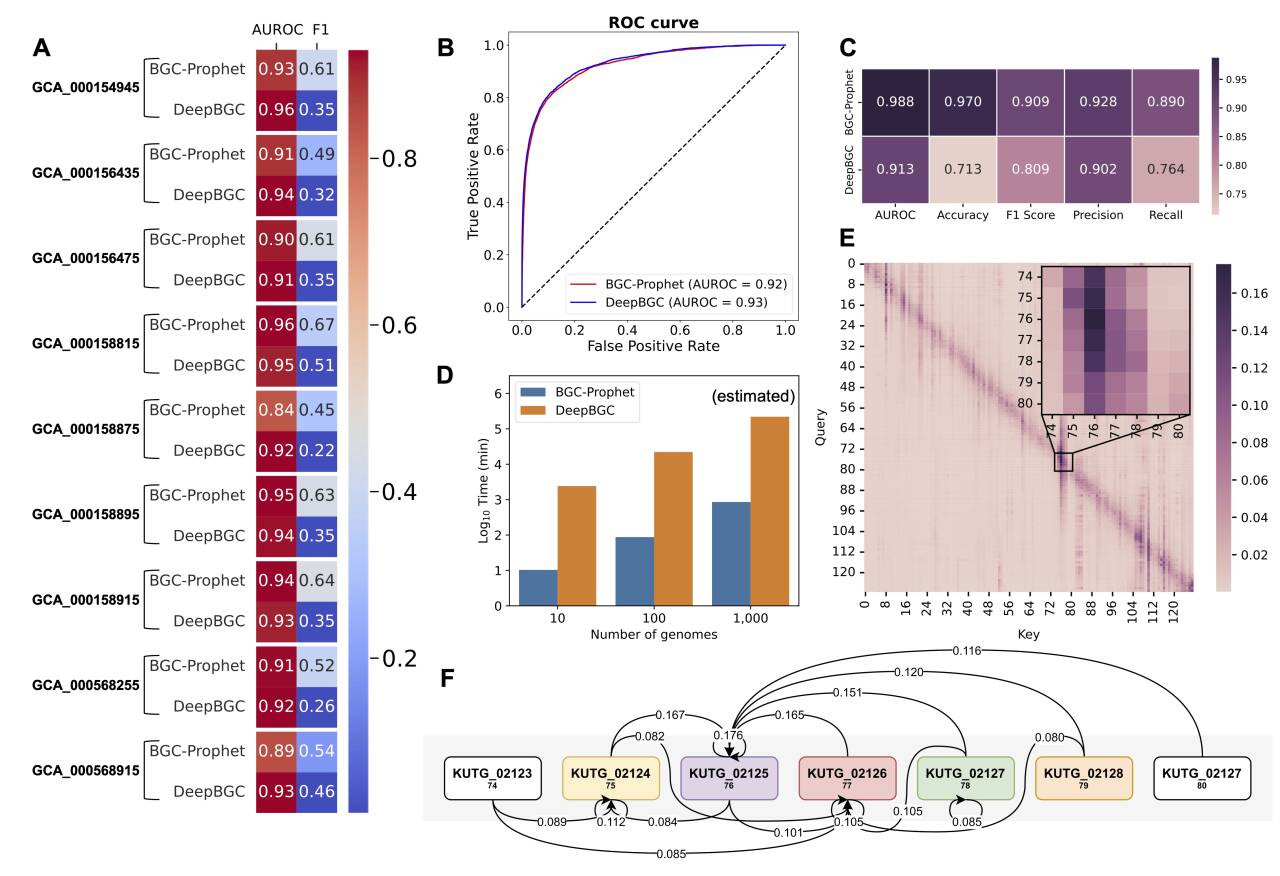
图1. BGC-Prophet可进行快速准确的BGC预测,并通过注意力机制揭示BGC中的核心基因
核心技术突破
1. 处理效率大幅提升
BGC-Prophet平均每分钟可完成一个基因组的BGC预测,速度是当前工具的240倍,支持大规模高通量分析。
2. 精准识别新型BGCs
BGC-Prophet在基准测试中AUROC值超过90%,显示出出色的预测准确性;且在曲霉属中,BGC-Prophet成功识别出超过16万个潜在BGCs,是现有方法的三倍。
3. 挖掘未知合成能力
BGC-Prophet能够识别现有分类体系之外的“未知类型”BGCs,在多个宏基因组样本中揭示了百万级潜在合成路径。
微生物视角下的地球生命演化史
团队还利用BGC-Prophet对大规模基因组数据进行分析,探索了微生物BGCs随地质事件的演化趋势:
24亿年前“大氧化事件”后,放线菌门中的聚酮类BGCs数量迅速增加,提升约3倍;
寒武纪大爆发时期,古菌中萜类BGCs多样性显著上升,增长约50%。
这些发现提示,环境剧变可能促进了微生物合成能力的跃迁,为生命演化提供了新视角。
应用前景广阔
BGC-Prophet的发布为合成生物学相关研究与产业转化提供了关键技术支撑:
药物研发:在海洋微生物中挖掘出3.2万种新型非核糖体肽(NRPs),为抗癌药物研发提供新的候选分子;
精准调控:通过注意力机制识别关键酶基因,提高工程菌改造效率;
环境应用:揭示甲烷氧化菌中具有特殊功能的BGCs,有望用于温室气体转化与生态修复。
开源共享,赋能科研社区
BGC-Prophet现已在GitHub平台开源,旨在服务全球科研人员,推动合成生物学技术普及与创新。论文第一作者赖奇龙表示:“我们希望通过AI模型,让更多科研人员能够高效解析微生物‘语言’,加速天然产物的发现与利用。
华中科技大学生命科学与技术学院本科生赖奇龙、姚帅、博士生查毓国为该论文共同第一作者,博士生张皓鸿等也参与了此工作,宁康教授、白虹正高级工程师为该论文共同通讯作者。
研究链接:
Nucleic Acids Research 原文链接:https://academic.oup.com/nar/article/53/7/gkaf305/8113170
GitHub 项目地址:https://github.com/NingLab-HUST/BGC-Prophet