2022年10月26日,生命学院、教育部分子生物物理重点实验室刘剑峰教授研究团队在Nature杂志子刊《自然·通讯》(Nature Communications)在线发表题为“Biased signaling due to oligomerization of the G protein-coupled platelet-activating factor receptor”(G蛋白偶联的血小板激活因子受体的偏向性信号起因于受体的寡聚化)的研究论文。

G蛋白偶联受体(GPCR)是细胞膜上具有七次跨膜螺旋(7TM)结构的受体家族,也是人体内最大的膜蛋白家族,广泛参与疾病的发生和发展,是重要的药物靶标。市场上1/3的药物靶向GPCR及其信号通路。GPCR包含A、B、C和F四种类型。其中,A类GPCR成员数量最多。除C类GPCR必须形成二聚体的发挥功能外,其他类GPCR以单体形式即可激活下游信号,同时也被发现能形成二聚体或寡聚体。但其聚合形式与生理功能和信号调控的相关性还并不清楚。GPCR的下游信号主要由G蛋白和β-arrestin介导。两种信号之间的平衡可以受到偏向性配体的调控。偏向性配体选择性激活某些信号,同时减少或阻止其它信号,为开发副作用小的药物提供了方向。但GPCR偏向性信号产生的内在机制还有待阐明。
血小板激活因子受体(Platelet-Activating-Factor Receptor,PAFR)属于A类GPCR,与血小板聚集和炎症反应密切相关。在内源配体PAF的刺激下,PAFR可以激活Gq和Gi蛋白,并募集β-arrestin引起受体内化。2017年,刘剑峰研究团队组参与了中科院吴蓓丽/赵强/张凯等解析的非激活态的PAFR晶体结构中的功能检测工作,验证了配体与PAFR的结合口袋(Nat. Struct. Mol. Biol.,2017),然而PARF如何激活下游不同信号通路还有待深入研究。在本研究项目中,刘剑峰研究团队采用PAFR作为A类GPCR的代表,通过单分子成像和生物发光共振能量转移技术,发现在低表达水平下,PAFR在细胞膜上即存在单体、二聚体和寡聚体的混合形式。采用该团队前期在C类GPCR二聚体研究中建立的基于SNAP的半胱氨酸交联技术(Nat Chem Biol., 2015; Nat Commun., 2019; PNAS., 2020),发现PAFR通过TM1、TM4和TM5相互作用形成二聚体,并且在激活过程中两个单体之间不发生的跨膜区构象重排,这与C类GPCR二聚体激活时的TM6和TM6相对的结构特征存在差异。接着,PAFR被锁定在二聚体和寡聚体时,PAF可以偏向性地引起更强的G蛋白信号激活,并减少对β-arrestin的募集以及相关的受体内化过程。同时,β-arrestin的募集通过减少PAFR二聚化和寡聚化状态,影响下游信号的偏向性(图1),这为GPCR偏向性信号调控提供了新机制,也为偏向性药物开发提供了新的方向。最后,在一种PAFR的天然遗传突变体中,他们发现二聚体和寡聚体比例增多,改变了G蛋白和β-arrestin信号的偏向性,这为GPCR存在不同聚合形式提供了生理意义。
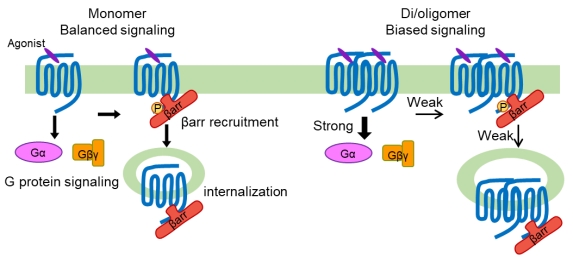
图1:GPCR通过二聚化和寡聚化产生偏向性信号的示意图
华中科技大学生命科学与技术学院博士后刘俊科和博士研究生汤恒敏为本研究论文的共同第一作者。华中科技大学刘剑峰教授、中科院生物物理研究所纪伟研究员以及法国功能基因组研究所J-P Pin和P Rondard教授为共同通讯作者。该研究受到科技部国家重点研发计划、国家自然科学基金以及教育部“111引智计划”的支持。
此项工作是刘剑峰团队对GPCR激活机制研究的又一重要发现。刘剑峰教授长期从事膜蛋白结构与功能相关研究,尤其对GPCR二聚体结构功能相关性、膜受体动态激活机制及下游信号转导通路等开展了系统深入的研究,先后以通讯作者在Cellx2、Nature x2、Nature Chemical Biology x2、Nature Communications x3、Cell Research x2、Science Advances、PNAS x2、Science Signaling、eLife x2等国际知名期刊上发表了一系列研究论文。此项研究展示了GPCR二聚体和寡聚体的作用新模式及对偏向性信号调控的新机制,拓展了GPCR的信号整合理论。
原文链接:https://doi.org/10.1038/s41467-022-34056-4