12月22日,国际权威学术期刊《生物信息学简报》(Briefings in Bioinformatics)在线发表了生命学院郭安源教授团队开发的癌症基因组大数据分析平台。该论文题目为《GSCA: an integrated platform for gene set cancer analysis at genomic, pharmacogenomic and immunogenomic levels》。郭安源教授及武汉科技大学曾燕教授为共同通讯作者,生命学院博士后柳纯洁、武汉科技大学胡斐斐和生命学院博士生谢贵燕为共同第一作者。
随着癌症基因组大数据的产生,准确分析和解读海量的组学数据成为新的挑战。针对这一需求,郭安源教授团队在2018年开发了多合一癌症基因集分析平台GSCALite(http://bioinfo.life.hust.edu.cn/web/GSCALite/),揭示癌症基因集的表达、突变、拷贝数变异和甲基化,及其与药物敏感性和临床特征的关联性,使得没有任何编程技能的实验生物学家也可使用癌症组学大数据,极大地方便了癌症研究。GSCALite自2018年发表在《生物信息学》(Bioinformatics)杂志以来,受到了广泛的使用和引用,截至目前已累积被使用了超过50万次,被引用了454次(谷歌学术),是Web of Science高被引论文。
癌症的发生发展通常是一组基因或者通路异常导致的,单个基因的异常信号可能会在背景噪声中淹没。针对基因集的整体分析可以提高信噪比,在基因集水平识别潜在的生物学信号。然而,目前缺乏使用基因集分析方法探索癌症多组学数据的平台。因此本研究将GSCALite升级为GSCA(http://bioinfo.life.hust.edu.cn/GSCA),其整合了33种癌症的基因组、药物基因组和免疫基因组数据。与GSCALite相比, GSCA着重体现基因集整体分析的思想,主要更新了以下三个特性: 1)基因集的整合表达水平(GSVA评分)与临床结局(生存和疾病TNM分期)的关联性分析;2)基因集的整合突变情况与4种生存时间的关联分析;3) 基因集的整合表达水平和整合突变情况与免疫细胞丰度的关联分析。下表通过与GSCALite及其他癌症多组学分析平台进行比较,展示GSCA的独特特征。

作者基于TCGA的癌症多组学数据、免疫丰度数据和小分子药物敏感性数据等,开发生物信息计算方法,构建癌症基因集分析平台GSCA。用户通过输入基因集或者通路中的基因名,选取分析类型和癌症类型,提交后即可获得能够用于发表的精美图片结果。GSCA是第一个利用基因集分析进行癌症多组学和免疫丰度分析及可视化的网页数据库平台。GSCA(http://bioinfo.life.hust.edu.cn/GSCA/)主要的功能模块包括:①基因表达与临床结果关联;②免疫细胞丰度与基因组特征关联分析;③基因突变与临床结果关联分析;④基因的药物敏感性分析。作者经过示例分析发现,利用GSCA的基因集分析方法,可以发现在单基因水平无法发现的临床新关联。
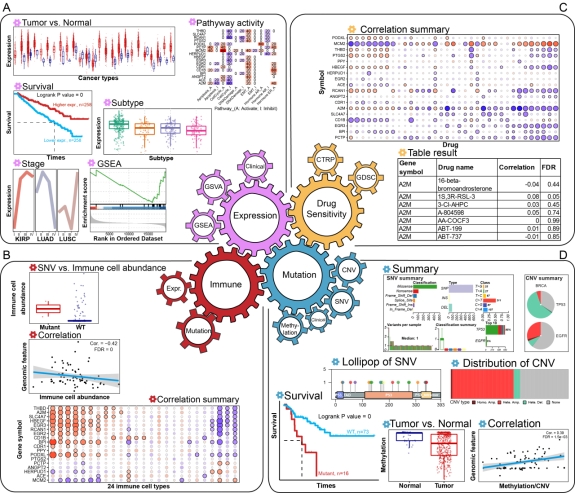
GSCA的原理图